海院科研动态(146)| 近海水体与表层沉积物中活跃、休眠及死亡微生物的季节性与生态位分化多样性
研究背景
应用16s/18s rRNA基因高通量测序技术已极大增进了我们对不同生态系统中微生物多样性及其群落组成时空动态的认识。然而,这些基于环境样本总DNA的宏条纹码技术,所捕获的既包含活跃的也混杂着休眠及死亡的微生物身份信息。因此,基于环境DNA的测序高估了实际参与生物地球化学循环的活跃微生物的多样性与丰度。如何高效地区分环境样品中活跃、休眠与死亡类群仍是当前面临的重要挑战。
近年来,本课题组将核酸染料叠氮溴化丙啶(PMA)处理技术与基于环境样品DNA/RNA的测序方法相结合,建立了三重宏条纹码测序与分析方法(Triple Metabarcoding Approach, TMA),成功区分了潮间带表层沉积物中活跃、休眠与死亡的微型真核生物,并追踪了其在室内干湿交替培养过程中生理状态与多样性的动态变化(图1)。然而,目前基于生理状态的细菌与古菌多样性尚未得到系统阐明,浮游生境中微生物生理状态与时空变化机制也仍需深入探究。基于上述科学问题,我们在受淡水影响的浅海沿岸区域开展夏冬两季采样,探讨表层、底层水体及表层沉积物中的细菌、古菌和微型真核生物的生理生态及其时空变化规律(图2)。
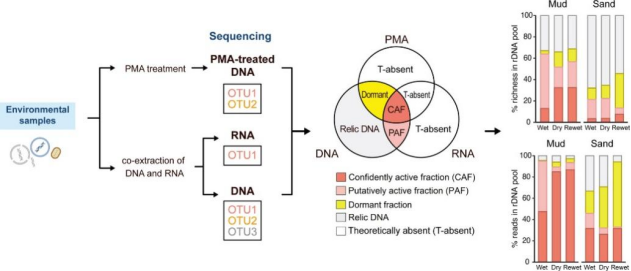
图1 三重宏条纹码测序与分析法 (Deng et al., 2024)
图2 本研究采样站位图
研究结果
对夏季和冬季水体及沉积物样本的总rDNA库进行分析时发现:休眠与死亡微生物的ASV数目占比超过半数(>50%),高于活跃微生物的占比(<50%)。尽管活跃微生物的ASV丰富度相对较低,其在rDNA库中的序列丰度却很高,占总体的50%-92%。其中,水体中活跃微生物的比例(平均85%)显著高于沉积物(52%)(图3)。
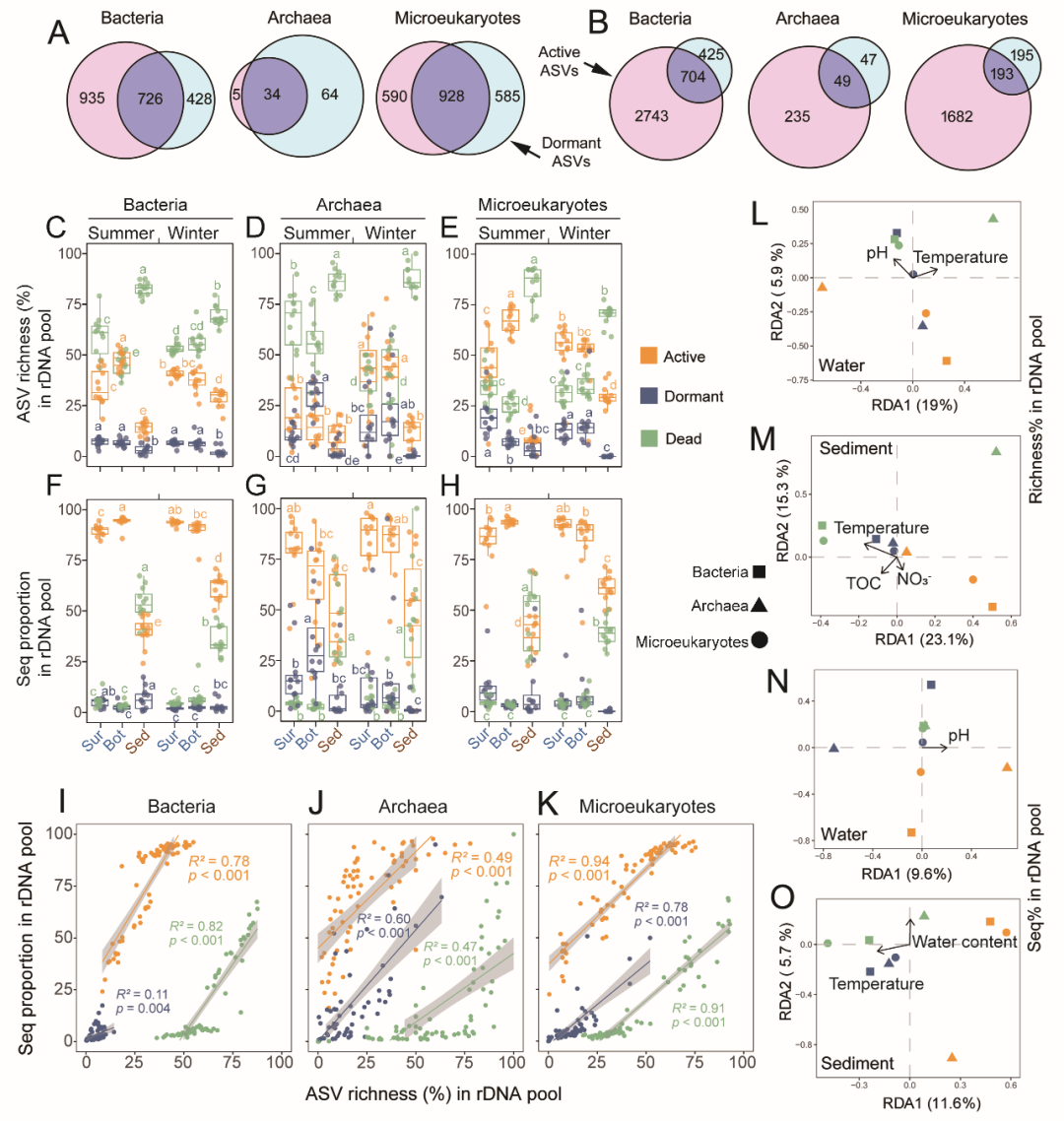
图3 活跃、休眠与死亡微生物ASV在水体与沉积物rDNA库中的分布及其环境驱动模式
不同生理状态的微生物群落组成明显不同。基于活跃微生物群落分析得到的ASV丰富度变化模式与基于总DNA测序数据所揭示的模式也存在显著差异(图4)。
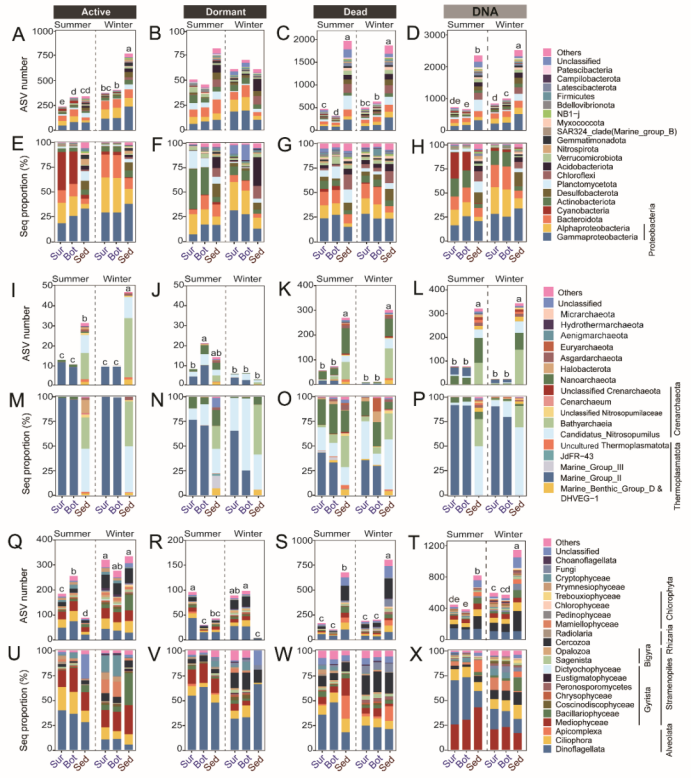
图4 活跃、休眠与死亡微生物以及总DNA测序所表征的微生物群落组成与多样性
水体与沉积物中活跃细菌、古菌和微型真核生物的ASV丰富度与特定的环境因子组合呈显著相关,而这些关联因子往往不同于基于总DNA得到的ASV丰富度的那些显著相关环境因子(图5)。
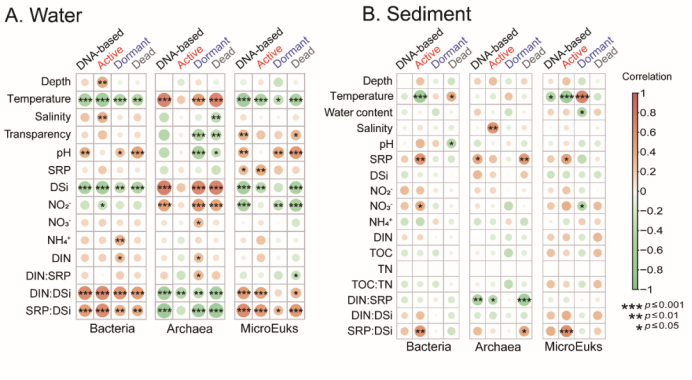
图5 环境因子与基于生理状态区分及基于总DNA的微生物群落多样性之间的相关性
在解释群落结构变异时,温度通常是水体与沉积物中所有活跃、休眠及死亡微生物包括基于总DNA表征的微生物群落结构的最强驱动因子。然而,驱动活跃微生物群落结构变异的显著相关的环境变量更多,特别是营养盐、pH和水体透明度,这些因子能解释更高比例的群落结构变异(图6)。
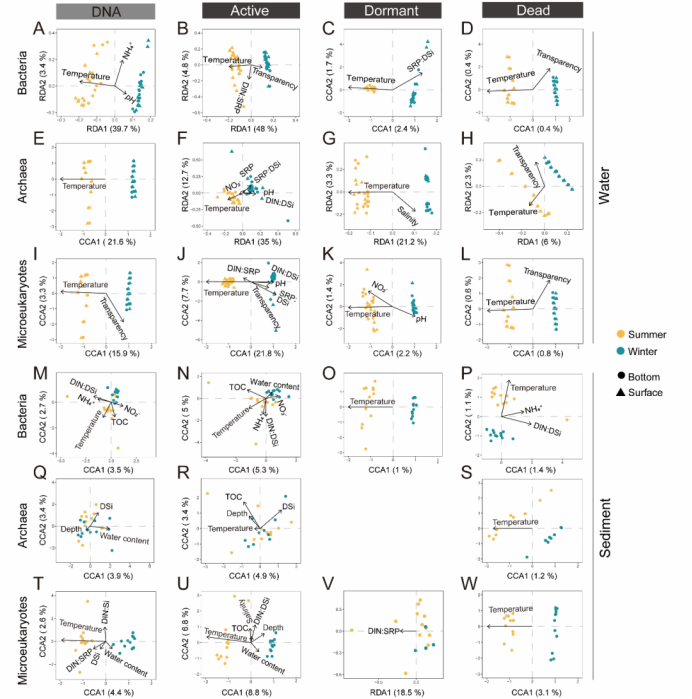
图6 解释不同生理状态微生物与总DNA表征微生物群落结构变异的关键环境因子比较
研究意义与总结
本研究采用三重宏条纹码测序与分析技术,揭示了活跃细菌、古菌、真核微生物群落对环境因子的响应模式与传统总DNA方法所得结果存在显著差异,提示仅凭DNA分析可能误判驱动微生物地球化学循环的关键因素。研究发现近海环境中存在的大量微生物类群均可能具有休眠状态,且呈现出显著的季节变化及生境分异模式。该成果展示了三重宏条纹码测序与分析技术在环境微生物分子生态学研究上的优势,凸显了在未来研究中整合生理状态解析的必要性,以准确揭示微生物的生态功能、动态变化与生物地理分布格局及其环境驱动机制。
文章相关信息
上述研究成果以“Seasonally and niche-differentiated diversity of active, dormant and dead microbes in coastal waters and surface sediments”为题,发表于 Nature Index收录期刊、中科院一区Top期刊《Communications Biology》,我院博士研究生李官哲与龚骏教授为论文共同通讯作者,生态环境部华南环境科学研究所余云军高级工程师为第一作者。本研究得到国家自然科学基金42376086、南方海洋科学与工程广东省实验室(珠海)创新团队项目(311021004)、广东省基础与应用基础研究基(2022A1515240020)等项目资助。
文章链接
https://www.nature.com/articles/s42003-025-08789-z



